上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
产品资料 – RNA 试剂 – cDNA 合成
模板置换反转录酶混合液 收藏
货 号
规 格
价 格(元)
北京库存
上海库存
广州库存
成都库存
苏州库存
武汉库存
#M0466L
100 reactions
4,789.00
有
有
有
无
有
无
#M0466S
20 reactions
1,199.00
无
有
有
无
无
无
Download:
- isoschizomers |
- compatible ends |
- single letter code
概述
模板置换反转录酶和其相应的反应缓冲液能够在反转录反应中有效地实现模板置换。混合液中含有独特的反转录酶和小鼠 RNase 抑制剂。与竞争对手的反转录酶产品不同,NEB 的产品不需要添加任何添加剂(如 PEG 或甜菜碱)来优化性能,从而简化了反应建立流程。与模板置换寡核苷酸(TSO)搭配使用, 合成的 cDNA 3’端可以选择性地加入已知序列。由此获得的 cDNA 可进行 PCR 扩增,也可作为 5’RACE( cDNA 末端快速扩增)或第二链 cDNA 合成的模板。
特性
以极低起始量模板(单细胞/细胞核或 2 pg 总 RNA)制备 RNA-seq 文库
以最低起始量为 10 ng 的总 RNA 进行 5’RACE
降低 RNA-seq 或 5’RACE 的背景
该酶混合液包含小鼠 RNase 抑制剂,不需要额外添加
兼容各种模板置换寡核苷酸(TSOs)、反转录引物和 DNA 聚合酶,用于扩增全长 cDNA
与其他的 RNA-seq 方法(例如:Smart-Seq)相比,操作更快捷
模板置换反转录酶混合液中的反转录酶(RT)在其到达 RNA 模板的 5’末端后会添加几个非模板源的核苷酸。这几个非模板源的核苷酸可以与已知序列的模板置换寡核苷酸(TSO)退火,促使反转录酶从 RNA 模板上切换到 TSO 上。得到的 cDNA 含有 3’末端添加有已知序列(TSO 的互补序列)。该特征可用于多种下游应用,例如 cDNA 扩增、5’RACE(cDNA 末端快速扩增)和第二链 cDNA 合成。说明书中有优化的实验流程。
图 1:模板置换流程概览
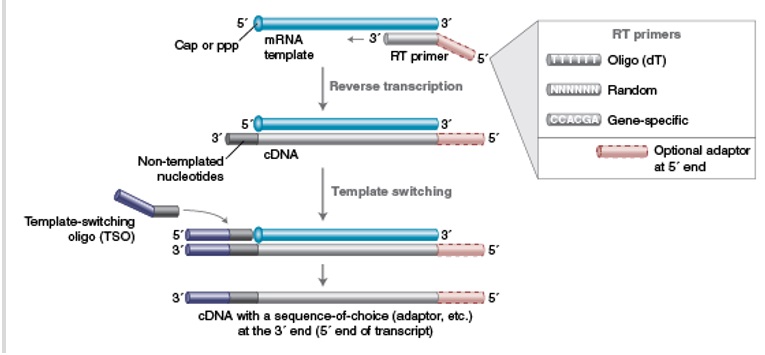
当到达 RNA 模板的 5’端时,反转录酶会在 cDNA 的 3’端添加几个非模板源的核苷酸。这些非模板源的核苷酸可以与包含已知序列的模板置换引物退火,从而促使反转录酶从 RNA 模板上切换到 TSO 上。所得到的 cDNA 在 3′ 端包含一个通用序列(TSO 的互补序列)
图 2:在单细胞 RNA-seq 实验中,模板置换反转录酶混合液相对于 Smart-seq2 方法的优势
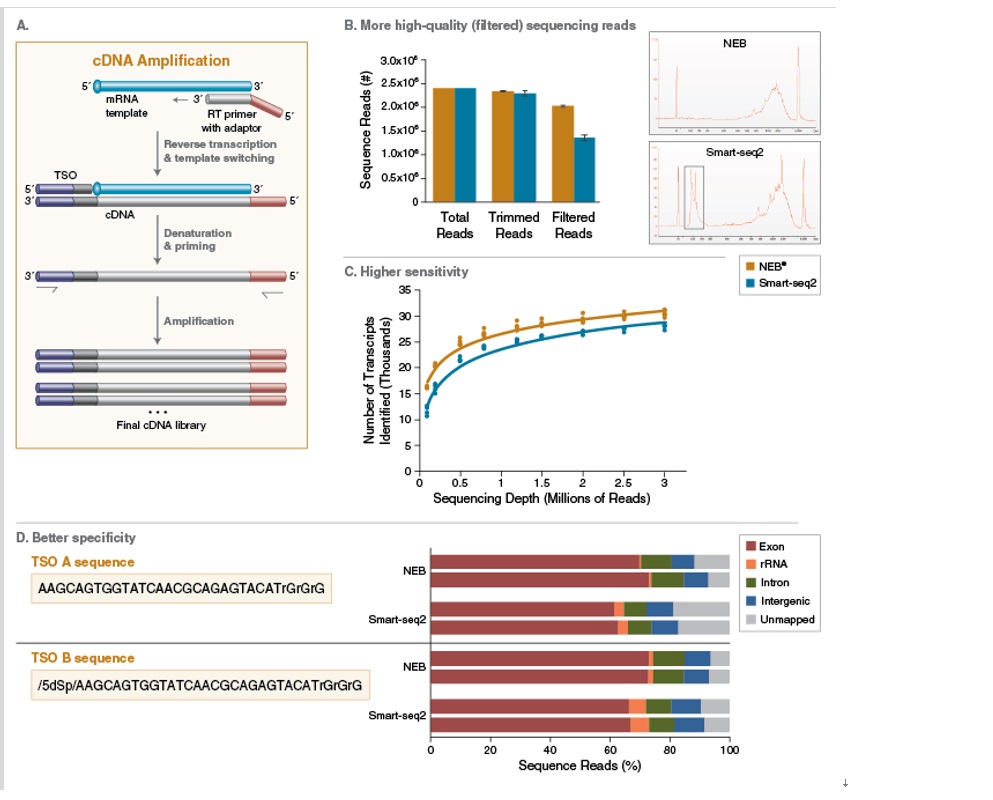
gA.模板置换介导的 cDNA 扩增概述。反转录引物含有 5’接头,它与 TSO 一起在 cDNA 的 5’和 3’末端添加接头。之后可用识别接头序列的引物 PCR 扩增全部反转录产物。
B-D.使用 NEB 模板置换反转录酶混合液或 Smart-seq2 方法(Picelli, S. et al.(2014). Nat. Protoc. 9, 171-81),用 10 μg Universal Human Reference(UHR) RNA (Agilent)与 ERCC RNA Spike-In Mix I(Thermo Fisher Scientific)生成 cDNA 文库。然后使用 NEBNext® Ultra™ II FS DNA 文库制备试剂盒(NEB#E7805)将每个 cDNA 文库制成 Illumina 文库,并在 Nextseq 500 上使用 2 x 75 个循环测序。将测序采样 2 x 1.2 M 个 reads(除非另有说明),去掉接头 reads,并且使用 Prinseq 过滤。B.显示的是样品总 reads(Total reads)、去掉接头的 reads(Trimmed reads)和通过 Prinseq 过滤的 reads(Filtered reads)。插图列举了每种方法制备的 cDNA 文库的 Agilent Bioanalyzer 2100 结果。虚线框标注了非特异片段。C.使用 Salmon 将过滤的 reads 与 GENCODE 28 和 ERCC 转录物比对。图中的点表示从每个文库检测到的 TPM(每百万转录数)≥1 的转录物的数量作为测序深度。 D.使用 Hisat 2.0.7 将过滤的读数与 hg19 人参考基因组比对,并使用 Picard SAM / BAM RNA Seq Metrics 工具计算 RNA-seq 指标。该图显示了分布于外显子(红色),rRNA(橙色),内含子(绿色),基因间区域(蓝色)的匹配 reads 和不匹配 reads(灰色)的百分比。
图 3:模板置换反转录酶混合液在 5’RACE 的实验中操作简单,性能优异
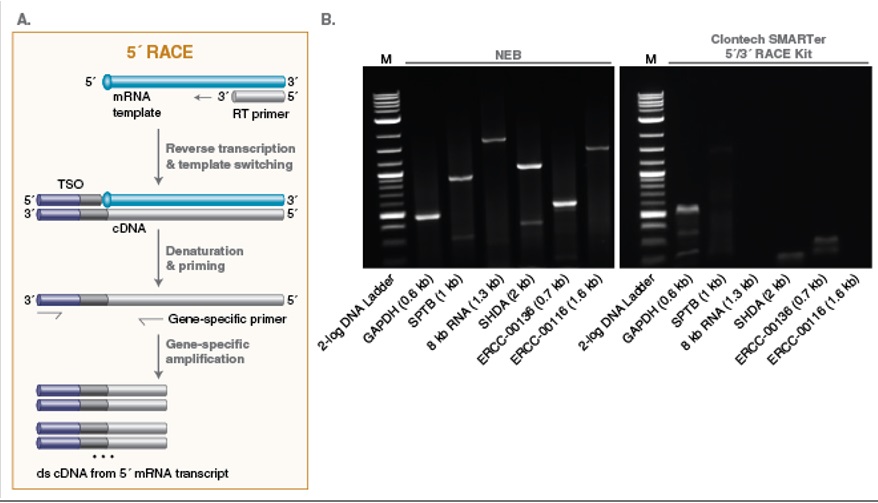
A.模板置换介导的 5’RACE 概述。在模板置换反转录反应后,用反向基因特异性引物和正向 TSO 特异性引物进行 5’RACE PCR。
B.使用 NEB 模板置换反转录酶混合液 5’RACE 方案(左)或 Clontech SMARTer 5’/3’RACE 试剂盒(右)对多种 RNA 靶标的 5’RACE 产物进行琼脂糖凝胶分析。加入 1 μg 的 Jurkat 总 RNA,10 pg 的 8 kb 合成 RNA 和 10 ng 的 ERCC RNA Mix 1,用于评估作为转录物长度和拷贝数的性能。在 NEB 的反应中,反转录引物为 oligo (dT)40 VN,TSO 为 GCTAATCATTGCAAGCAGTGGTATCAACGCAGAGTACATrGrGrG,TSO 中特异性 PCR 引物用下划线标注。两种方法使用相同的基因特异性 PCR 引物。靶标名称和预期片段长度如图所示。
图 4:模板置换反转录酶混合液在第二链 cDNA 合成中操作简单,且可捕获完整的 5’末端转录产物

A.模板置换介导的第二链 cDNA 合成概述。在反转录反应后,RNA 模板水解,使用 TSO 作为引物延伸合成第二链 cDNA。
B.使用 1 kb 的合成 RNA 作为模板,使用 poly(dT)40 VN 作为第一链 cDNA 合成的反转录引物。使用模板置换介导的方法或 Gubler 在文献中描述的方法—R and Hoffman,BJ.(1983) Gene,25,263-269,对第二链 cDNA 合成进行三个平行实验。所得到的双链 cDNA 产物使用反向引物用 Sanger 法对第二链 cDNA 进行测序。结果显示模板含有转录起始位点(TSS),并标注出了匹配序列。 添加到 cDNA 末端的 TSO 序列用灰色标注。未通过 Gubler 和 Hoffman 方法检测到的序列用蓝色标注。不可信读序区用黄色标注。
随产品提供的试剂
|
储存温度(℃) |
浓度 |
|
|
模板置换反转录缓冲液 |
-20 |
4 X |
产品类别:
cDNA合成和反转录产品
PCR,qPCR 和 扩增技术产品
应用
cDNA 合成
RNA 分析
储存温度
-20°C
